ECGAN
Erzeugung und Detektion von Anomalien in Zeitreihen mit Hilfe von Generative-Adversarial-Networks
Fortschritte in der maschinengestützten Auswertung von medizinischen Daten können dabei helfen, medizinisches Fachpersonal zu entlasten und die medizinische Versorgung durch einen effektiveren und effizienteren Einsatz von Ressourcen zu unterstützen. Dies gilt so auch für Elektrokardiogramme (EKGs), mit Hilfe derer viele Herz-Kreislauf-Erkrankungen diagnostiziert werden und bei denen die Erkennung von anormalen Umständen Leben retten kann.
Seit einigen Jahren werden für eine autonome Detektion von Anomalien vermehrt Methoden des maschinellen Lernens eingesetzt. Im Rahmen des Forschungsprojektes ECGAN wurde hierbei eine neue Herangehensweise zur Generierung von EKG-Daten und zur Detektion von Anomalien basierend auf Generative Adversarial Networks (GANs) entwickelt. GANs sind eine spezielle Klasse neuronaler Netze, mit Hilfe derer Verteilungen von Daten erlernt werden können. Hierbei erzeugt ein generierendes neuronales Netz (der Generator) möglichst realistische synthetische Daten und ein diskriminierendes Netzwerk (der Diskriminator) soll diese von realen Daten unterscheiden. Das Training von GANs ist jedoch sehr instabil und viele bestehende Ansätze zur Detektion von Anomalien sind zu ressourcenintensiv für eine Echtzeitanalyse.
Ziel des Projekts ECGAN war daher die Stabilisierung des Trainings von GANs zur Generierung von EKG-Daten und eine Beschleunigung und Verbesserung der Anomaliedetektion. Hierfür wurden zunächst bestehende GAN-basierte Ansätze verbessert. Zusätzlich wurde der klassische Generator durch einen β-Variational-Autoencoder ersetzt. Dies führte zu der erhofften Stabilisierung des Trainings und einer Erhöhung der Qualität der generierten Daten im Vergleich zu Vorarbeiten. Weitere Verbesserungen führten zu einer Beschleunigung der Anomaliedetektion, wodurch nun eine Echtzeit-Anomaliedetektion ermöglicht wird. Zudem konnte die Anomaliedetektion durch die gezieltere Nutzung der GAN-Komponenten verbessert werden.
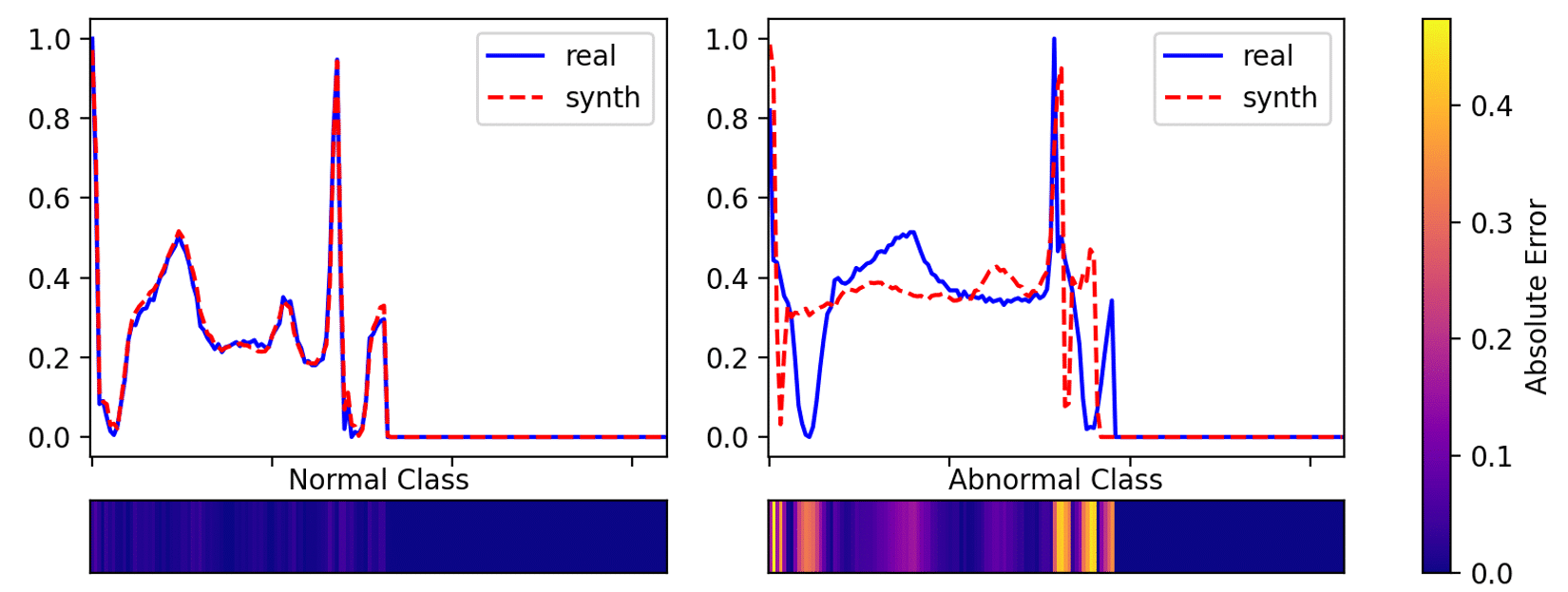
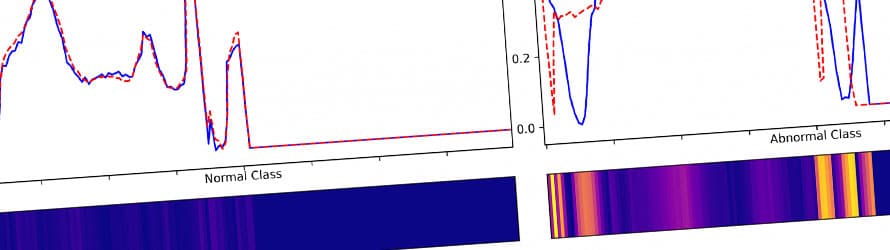
Vergleich eines realen Herzschlags (blau) mit der Rekonstruktion (rot). Die Farbintensität der Heatmap stellt den Abstand zwischen dem realen und synthetischen Herzschlag dar. Der normale (healthy) Herzschlag kann detailliert rekonstruiert werden, der anormale Herzschlag ist wie zu erwarten nicht durch das Model rekonstruierbar, da das Netzwerk nur auf Daten der normalen Klasse trainiert wird. (Quelle: eMundo GmbH)
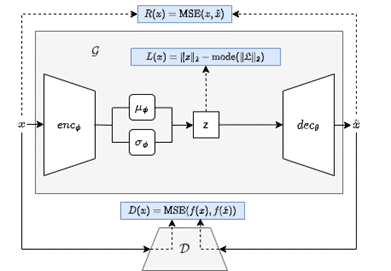
Architektur des entwickelten β-VAEGAN Modells (Quelle: eMundo GmbH)
Fortschritte in der autonomen Anomaliedetektion profitieren von der offenen Forschung. Die offene Forschung ist nicht nur für die Reproduzierbarkeit selbst wichtig, sondern ebenfalls für eine schnelle und gezielte Weiterentwicklung und Vergleichbarkeit von Methoden. Aus diesem Grund wurden die Ergebnisse im Rahmen eines Frameworks für die reproduzierbare Verarbeitung und Analyse von EKG-Daten auf GitHub veröffentlicht. Durch die hohe Modularität des Frameworks ist es zudem möglich, auch andere Zeitreihen leicht zu generieren, wie beispielsweise EEG-Daten, und Anomalien in den Daten zu erkennen.
„Die Zusammenarbeit mit der BayFIA war exzellent und ist sehr empfehlenswert. Mit Hilfe fachlich kompetenter Ansprechpartner konnte eine initial nur grobe Idee schnell und wertschätzend konkretisiert werden. Während sämtlicher Projektstufen konnten wir uns über ein großes Interesse sowie zeitnahe Hilfe freuen und fokussiert forschen. Vielen Dank!“
Fiete Lüer, eMundo GmbH
Von der Idee über das Forschungsprojekt in die offene Forschung
Die eMundo GmbH hat gemeinsam mit der LMU München im Januar 2020 Kontakt mit der Bayerischen Forschungsallianz aufgenommen, um Fördermöglichkeiten für ihre Projektidee zu prüfen. Im Rahmen einer individuellen KMU-Beratung zu verschiedenen Förderprogrammen auf Landes-, Bundes- und EU-Ebene wurde die Bayerische Forschungsstiftung als potentielle Förderinstitution identifiziert und daher die Projektskizze mit Einverständnis der Konsortialpartner an diese weitergereicht.
Nach Beratung und erfolgreicher Antragstellung bei der Forschungsstiftung konnten die Projektpartner ihr Vorhaben ECGAN von Mai 2020 bis Juli 2021 als Kleinprojekt mit einer Förderung i. H. v. 50.000 Euro erfolgreich durchführen. Die Ergebnisse wurden bereits kurz nach Projektabschluss auf internationalen Konferenzen und in wissenschaftlichen Magazinen veröffentlicht und der Quellcode in Form eines generalisierten Open-Source-Projektes öffentlich zugänglich gemacht.
Projektpartner


Ludwig-Maximilians-Universität München, Institut für Informatik,
Forschungsgruppe Data Mining in der Medizin

